中国农业科学院博士后开发新的基因型检测技术TAIL-peq
近日,中国农业科学院深圳农业基因组研究所(岭南现代农业科学与技术广东省实验室深圳分中心)常玉晓课题组联合钱前院士团队等在Plant Communications上发表了题为“Streamlined whole-genome genotyping through NGS-enhanced thermal asymmetric interlaced (TAIL)-PCR”的研究论文。该研究开发了一种基于非特异性扩增进行全基因组基因型检测(WGG)的高通量测序技术TAIL-PCR-seq (TAIL-peq)(图1),该技术具备重复性好、准确度高、成本低以及适用性广等特点,预期将在作物基因组育种中发挥重要作用。
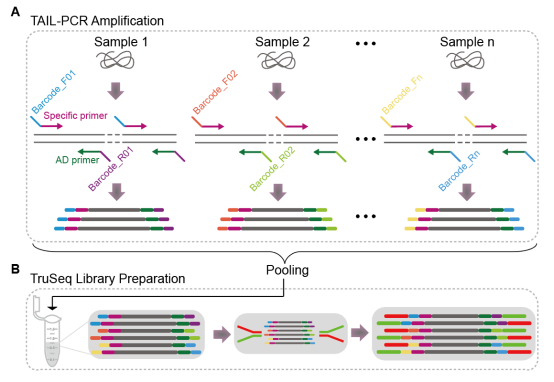
图1 TAIL-peq技术流程图
WGG是作物基因组育种中的一个重要环节。然而,目前基于高通量测序的WGG技术,仍然存在文库制备流程繁琐以及成本高昂等缺点,阻碍了此类技术在作物(尤其是小宗作物)分子育种中的应用。本研究在课题组前期开发的可用于同时进行前景基因筛选与遗传背景检测的FBI-seq技术(Zhao et al., JIPB, 2023)的基础上,借鉴热不对称交错PCR (Thermal Asymmetric Interlaced PCR,简称TAIL-PCR)技术,通过利用AD简并引物替代之前Tn5转座酶所加的通用引物,并与SP特异引物进行配对,借助优化后的PCR扩增条件,使得SP与AD引物组合能在任意物种的基因组上进行稳定的非特异性扩增,进而获得数以万计、均匀分布整个基因组的tag标签位点以及SNP标记用于WGG。该研究首先对TAIL-peq的技术特征(包括可行性、重复性以及准确性等)进行了全面的评价(图2),随后证明了SP与AD引物均可对非特异性扩增产生重要影响,接着将TAIL-peq技术成功应用于一个玉米自然群体的群体结构分析以及水稻RIL群体的连锁图谱构建的研究中,最后证明了基于非特异性扩增,相同的SP和AD引物组合可以在大豆、小麦、高粱、辣椒等10种不同植物,以及猪和犬两种动物中通用,均表现出良好的WGG效果(图3)。
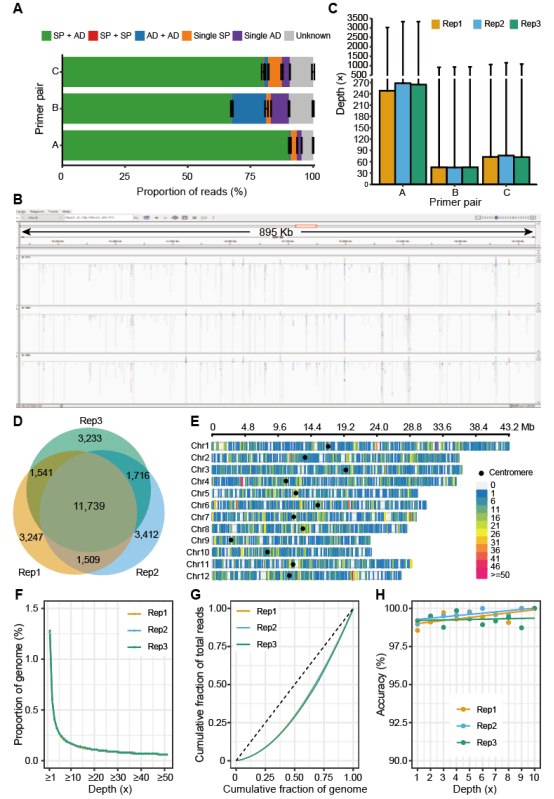
图2 TAIL-peq数据特征及技术评价
与全基因组重测序、简化基因组测序以及固/液相芯片等基因型检测技术相比,新开发的TAIL-peq技术无需基因组断裂的过程,仅需要一次非特异性的PCR扩增即可稳定获得数以万计、甚至十万级别的SNP变异位点用于WGG,而且单个样品的文库制备成本仅相当于一次PCR!预期TAIL-peq测序技术将在动植物的基因组育种中具备重要的应用价值,助力目标性状的改良及新品种的培育。
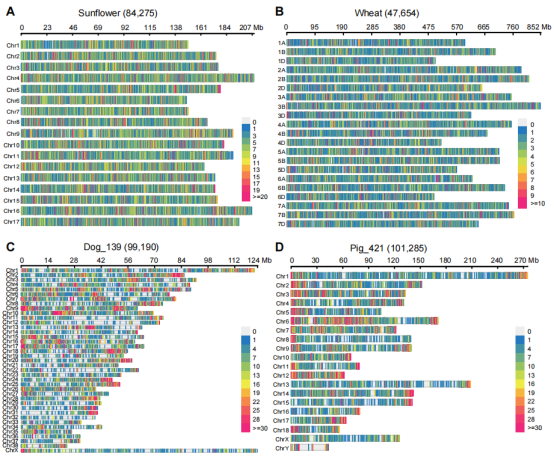
图3 TAIL-peq在动植物基因型检测中的应用
中国农业科学院深圳农业基因组研究所常玉晓研究员和钱前院士为本论文的通讯作者,博士后赵胜为论文的第一作者。基因组所向勇研究员、卢洪研究员、刘毓文研究员、博士生陈鹏、硕士生王悦、科研助理竺正航,以及广东省农科院柳武革研究员和王重荣研究员也参与了研究工作。本研究得到了广东省重点研发项目、国家自然科学基金(32300340, 32172086)、以及深圳市可持续发展项目的资助。

